antu
NGBler
Rosetta@home ist ein Projekt mit dem Ziel, die dreidimensionale Struktur von Proteinen und Proteinkomplexen vorherzusagen und gezielt zu erzeugen. Die Struktur eines Proteins bestimmt sich durch eine Abfolge von Aminosäuren, entlang derer sich das Protein "konstruiert". Man bezeichnet diesen Prozess auch als Proteinfaltung, dabei handelt es sich um eines der ältesten noch offenen Probleme in der Molekularbiologie. Es ist zwar möglich die Struktur von Proteinen experimentell festzustellen, allerdings ist das extrem teuer, und funktioniert auch nicht bei allen Proteinen. Deswegen wird eifrig an rechnerischen Methoden zur Bestimmung der Proteinstruktur geforscht. Die Forscher versprechen sich dadurch, die Funktionsweise von Proteinen besser zu verstehen und außerdem völlig neue Medikamente gegen schwere Krankheiten wie HIV, Malaria, Krebs oder Alzheimer zu finden.
Video über Rosetta@home und Proteine (Englisch):
Rosetta@home | � | ||
| Ziel | Vorhersage von Proteinstrukturen | � | |
| Bereich | Biochemie | Status | produktiv |
| Betreiber | Universität von Washington | Land | Vereinigte Staaten von Amerika |
| Beginn | 16. September 2005 | Ende | läuft noch |
| Betriebssysteme | Windows (32/64 bit), Linux (32/64 bit), OS X (Intel) | Kommerziell? | nein |
| Projekt-Webseite | Rosetta@home | Projekt-Forum | Rosetta@home Forum |
| Server-Status | Rosetta@home Server-Status | Benutzerkonto | Konto bei Rosetta@home |
| Zertifikat | Certificate of Computation | Abzeichen | nein |
| Arbeitspakete | Rosetta Mini, Rosetta | � | |
ngb:setiteam | � | ||
| Teamseite | ngb:setiteam bei Rosetta@home | Teambeitritt | ngb:setiteam beitreten |
| Team-Statistik | BOINCstats | Mitgliederliste | BOINCstats, Bakerlab |
Die Forschung
Proteine sind aus Aminosäuren (chemische Grundbausteine) aufgebaute Moleküle, die im menschlichen Körper viele wichtige Funktionen erfüllen. Die Struktur bzw. die Form eines Proteins bestimmt sich durch die Reihenfolge der enthaltenen Aminosäuren. Die Reihenfolge der Aminosäuren, also der "Bauplan" des Proteins, ist in einem Gen gespeichert.
Welche Funktion genau ein Protein im menschlichen Körper erfüllt wird durch seine Form bestimmt. Ein Beispiel: Ein Protein das Zucker spaltet, um einer Körperzelle die im Zucker gespeicherte Energie zu liefern, ist so geformt, dass es sich an den Zucker andocken kann (wie ein Schlüssel und ein Schloss). Sobald ein Zuckermolekül an das Protein angedockt ist, wird eine chemische Reaktion gestartet die den Zucker aufspaltet und der Zelle neue Energie liefert.
Die Struktur/Form eines Proteins lässt sich entweder durch Kristallstrukturanalyse oder Kernspinresonanz bestimmen. Diese Methoden sind aber sehr aufwendig, oft dauert es Wochen/Monate um ein einzelnes Protein zu analysieren, hinzu kommen hohe Kosten von bis zu 70.000 € pro Protein. Außerdem sind diese Methoden fehleranfällig und funktionieren nicht bei allen Proteinen. Deswegen ist von den über 35 Millionen verschiedenen Proteinen die bisher entdeckt wurden, nur von weniger als 100.000 die Struktur bekannt.
Daher suchen Forscher nach Algorithmen, mithilfe derer sich die Struktur eines Proteins rechnerisch aus der Reihenfolge der Aminosäuren (dem "Bauplan") errechnen lässt. Dabei machen sie sich zu nutze, dass Proteine in der Natur immer die Form annehmen, die den niedrigsten Energieaufwand hat. Das Problem dabei ist, dass die Anzahl der möglichen Formen die ein Protein annehmen kann, exponentiell mit der Anzahl der enthaltenen Aminosäuren steigt. Proteine im menschlichen Körper bestehen aus hunderten bis tausenden von Aminosäuren, dadurch gibt es viel zu viele Möglichkeiten um sie alle durch zu probieren.
Die Forscher haben daher Methoden entwickelt um den Suchraum stark einzuschränken. Diese Methoden basieren auf dem Wissen über die chemischen und physikalischen Wechselwirkungen zwischen den Aminosäuren und Kenntnissen darüber wie Aminosäuren sich üblicherweise anordnen.
Trotzdem müssen viele verschiedene Strukturen durchprobiert werden, bis die mit dem niedrigsten Energieaufwand gefunden ist. Und genau das macht Rosetta@home, es probiert viele verschiedene Strukturen aus, und die Struktur mit dem niedrigsten Energieaufwand ist dann die tatsächliche Struktur des Proteins, oder zumindest sehr nah dran. Dabei werden die Methoden zur Vorhersage der Struktur von den Forschern immer weiter verbessert, mit dem Ziel, irgendwann die Struktur von Proteinen schnell und exakt vorhersagen zu können. Wenn dieses Ziel erreicht ist, kann aus der errechneten Struktur auch auf die Funktion des Proteins geschlossen werden.
Damit wäre es dann auch möglich künstliche Proteine mit bestimmten Funktionen herzustellen, das bezeichnet man auch als "Proteindesign". Damit ließen sich bessere Medikamente/Impfstoffe herstellen und möglicherweise auch Heilmittel für Krankheiten wie HIV, Krebs oder Alzheimer finden.
Die Forschung von Rosetta@home ist ziemlich erfolgreich. Beim letzten CASP-Wettbewerb (dabei werden verschiedene Methoden zur Proteinstrukturvorhersage verglichen) hat das Rosetta@home-Team ziemlich gut abgeschnitten.
Informationen über Rosetta@home und Proteine:
Wer gerne mehr erfahren möchte über das Projekt und die Forschung rund um Proteine/Proteinfaltung, findet hier ein paar Links.
- Wikipedia-Seite zu Rosetta@home
- Was ist Rosetta@home und welchen Zweck haben die Vorhersagen von Proteinen und Proteinkomplexen?
- Das Problem der Proteinfaltung
- Wie Rosetta@home dabei hilft Krebs, Alzheimer, HIV und andere Krankheiten zu heilen.
- Oft gestellte Fragen über Rosetta@home.
- Video über Proteine und Proteinforschung in München (Deutsch)
- Tagebuch von David Baker über Rosetta@home
- Nachrichten und Artikel rund um Rosetta@home (Englisch)
- Wissenschaftliche Publikationen/Ergebnisse der Rosetta@home-Forschung
- Rosetta@home Twitter
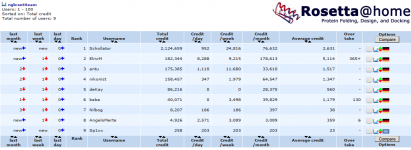
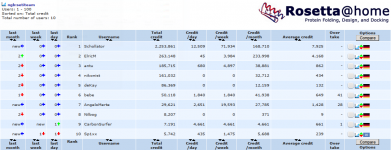
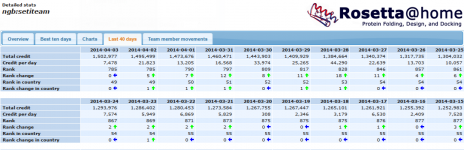
Statistik
Stand: 15.09.2020
| Mitglieder | 60 (29 aktiv) |
| Platzierung (nach Punkten, weltweit) | 146 |
| Platzierung (nach aktuellen Ø Punkten, weltweit) | 32 |
| Platzierung (nach Punkten, Deutschland) | 6 |
| Platzierung (nach aktuellen Ø Punkten, Deutschland) | 3 |
Meilensteine
- Am 15.01.2014 haben wir Platz 100 in Deutschland erreicht.
- Am 29.01.2014 haben wir Platz 75 in Deutschland erreicht.
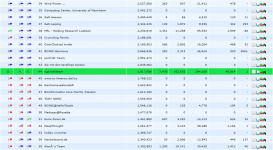
Wettbewerbe
Unser Team nimmt natürlich regelmäßig an Wettbewerben teil, um uns mit den anderen Teams zu messen.
- "Despite my age I don't live in the past, but in the future" 1st Rita Levi-Montalcini memorial: 15. Platz, 250.544 Punkte, 5 Teilnehmer.
- Bettina von Arnim Birthday-Challenge vom 04.04 - 06.04.2014: Platz 3/23, 645.789 Punkte.
- Florence Nightingale – Race: Platz 10/13, 1.782.321 Punkte.
Tipps/Hinweise
- Rechenzeit von Arbeitspaketen einstellen
Bei Rosetta@home gibt es eine Besonderheit, was die Berechnungsdauer eines Arbeitspakets angeht. Die Dauer lässt sich nämlich in den Projekt-Einstellungen festlegen, Rosetta@home schickt einem dann Arbeitspakete die ungefähr so lange zur Berechnung brauchen wie man es eingestellt hat. Das ist praktisch, wenn man Rechner nur sporadisch/kurz laufen lässt (kurze Arbeitspakete) oder wenn man z.B. weiß das der Rechner sowieso 24/7 läuft (lange Arbeitspakete). - Was tun, wenn Show Graphics keine 3D-Grafiken/Daten anzeigt? (Linux)
Ich dachte mir wir können dieses Thema nutzen um Tipps/Neuigkeiten rund um das Projekt zu sammeln/diskutieren, etc.
Änderungsprotokoll:
09.01.2014: "1st Rita Levi-Montalcini memorial" Wettbewerb hinzugefügt.
11.01.2014: Tabelle oben eingefügt. Kleine Umstrukturierungen. Beschreibung der Forschung von Rosetta@home hinzugefügt.
15.01.2014: Erster Meilenstein: Platz 100 in Deutschland!
29.01.2014: Bettina von Arnim Wettbewerb hinzugefügt, Platz 75 in Deutschland.
15.09.2020: Tabelle aktualisiert und Florence-Nightingale Wettbewerb hinzugefügt.
20.09.2020: Tipps/Hinweise aktualisiert.
11.01.2014: Tabelle oben eingefügt. Kleine Umstrukturierungen. Beschreibung der Forschung von Rosetta@home hinzugefügt.
15.01.2014: Erster Meilenstein: Platz 100 in Deutschland!
29.01.2014: Bettina von Arnim Wettbewerb hinzugefügt, Platz 75 in Deutschland.
15.09.2020: Tabelle aktualisiert und Florence-Nightingale Wettbewerb hinzugefügt.
20.09.2020: Tipps/Hinweise aktualisiert.
Zuletzt bearbeitet:











 Ich war vielleicht eine Zeit lang ein bisschen inaktiv hier im Forum, aber jetzt bin ich ja wieder da.
Ich war vielleicht eine Zeit lang ein bisschen inaktiv hier im Forum, aber jetzt bin ich ja wieder da. 

 Vielleicht hilft es wenn du den Leuten erklärst wie man mit R@h die Forschung unterstützt, was für Erfolge es da bereits gab und was für Möglichkeiten die Forscher noch für die Zukunft sehen.
Vielleicht hilft es wenn du den Leuten erklärst wie man mit R@h die Forschung unterstützt, was für Erfolge es da bereits gab und was für Möglichkeiten die Forscher noch für die Zukunft sehen. Die könnten laut dem Artikel in der Krebsforschung nützlich sein.
Die könnten laut dem Artikel in der Krebsforschung nützlich sein.